18 de Julho de 2024
Ao longo da história da humanidade, alguns eventos são vistos como marcos decisivos, provocando um impacto irreversível. Com as grandes áreas da Ciência não seria diferente. No início da década de 80, o pesquisador Kary Mullis desenvolveu uma técnica que viria a revolucionar a ciência: A descoberta da técnica da PCR – Polymerase Chain Reaction, ou Reação em Cadeia da Polimerase, foi publicada em algumas revistas científicas* a partir de 1985, e desde então abriu caminho para novas pesquisas e aplicações relacionadas ao estudo de DNA. Posteriormente, em 1993, essa descoberta rendeu a Kary Mullis o Nobel de Química, consolidando-o como referência no assunto.
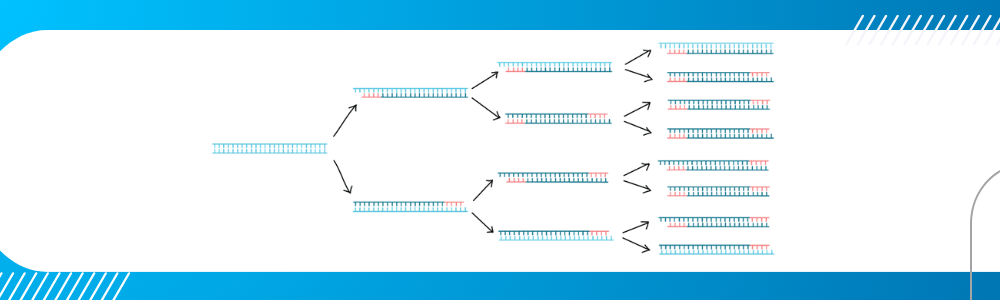
A PCR é uma técnica na qual se produz rapidamente milhões de cópias de um fragmento específico de DNA. Essa técnica foi revolucionária uma vez que o genoma de qualquer organismo está armazenado nas moléculas de DNA no interior das células, porém, para a análise desta informação genética, é necessária uma grande quantidade de DNA. Sendo assim, antes da descoberta da PCR, qualquer estudo relacionado a informações genéticas era repleto de limitações.
Com a PCR, a partir do uso de pequenos fragmentos de DNA sintético, chamados de primers, é possível selecionar um segmento do genoma que se pretende amplificar. Em seguida, a partir de vários ciclos da síntese deste fragmento de DNA, ele é amplificado.
Durante a reação de PCR, quem tem a capacidade de sintetizar uma nova fita de DNA complementar a partir da fita molde é a enzima DNA polimerase. A DNA polimerase adiciona o primeiro nucleotídeo por meio de um iniciador, que é o primer, o que permite delinear uma região específica da sequência modelo que se deseja amplificar. Ao final da reação de PCR a sequência específica estará acumulada em bilhões de cópias, os chamados amplicons.
 Para a realização da PCR, alguns componentes obrigatórios devem integrar a reação:
DNA Template: Se trata do modelo de DNA, ou a amostra de DNA que contém a sequência alvo.
DNA Polymerase: Enzima que sintetiza novas fitas de DNA, complementares à sequência alvo. É uma enzima altamente resistente ao calor.
Primers: Sequências curtas de DNA de fita simples que são complementares à sequência alvo. A polimerase começa a sintetizar a nova fita de DNA a partir do final do primer.
Nucleotídeos, ou desoxinucleotídeos trifosfatos (dNTPs): São, essencialmente, “blocos de construção” para novas cadeias de DNA. Cada dNTP é composto por uma base nitrogenada, um açúcar e um grupos fosfato. As moléculas de DNA contêm quatro diferentes bases nitrogenadas que compõem um desoxinucleotídeo trifosfato: Adenina (A), Timina (T), Citosina (C) e Guanina (G). Já as moléculas de RNA são compostas pelas bases nitrogenadas: Adenina (A), Uracila (U), Citosina (C) e Guanina (G). São as bases nitrogenadas, presentes nos nucleotídeos, que codificam a informação genética, uma vez que é a sequência de bases ao longo da molécula de DNA e RNA que determina a informação genética.
Cloreto de magnésio (MgCl₂): Atua como cofator essencial da atividade da DNA Polimerase, além de influenciar na estabilização dos nucleotídeos e na especificidade da amplificação do alvo na reação.
Para a realização da PCR, alguns componentes obrigatórios devem integrar a reação:
DNA Template: Se trata do modelo de DNA, ou a amostra de DNA que contém a sequência alvo.
DNA Polymerase: Enzima que sintetiza novas fitas de DNA, complementares à sequência alvo. É uma enzima altamente resistente ao calor.
Primers: Sequências curtas de DNA de fita simples que são complementares à sequência alvo. A polimerase começa a sintetizar a nova fita de DNA a partir do final do primer.
Nucleotídeos, ou desoxinucleotídeos trifosfatos (dNTPs): São, essencialmente, “blocos de construção” para novas cadeias de DNA. Cada dNTP é composto por uma base nitrogenada, um açúcar e um grupos fosfato. As moléculas de DNA contêm quatro diferentes bases nitrogenadas que compõem um desoxinucleotídeo trifosfato: Adenina (A), Timina (T), Citosina (C) e Guanina (G). Já as moléculas de RNA são compostas pelas bases nitrogenadas: Adenina (A), Uracila (U), Citosina (C) e Guanina (G). São as bases nitrogenadas, presentes nos nucleotídeos, que codificam a informação genética, uma vez que é a sequência de bases ao longo da molécula de DNA e RNA que determina a informação genética.
Cloreto de magnésio (MgCl₂): Atua como cofator essencial da atividade da DNA Polimerase, além de influenciar na estabilização dos nucleotídeos e na especificidade da amplificação do alvo na reação.
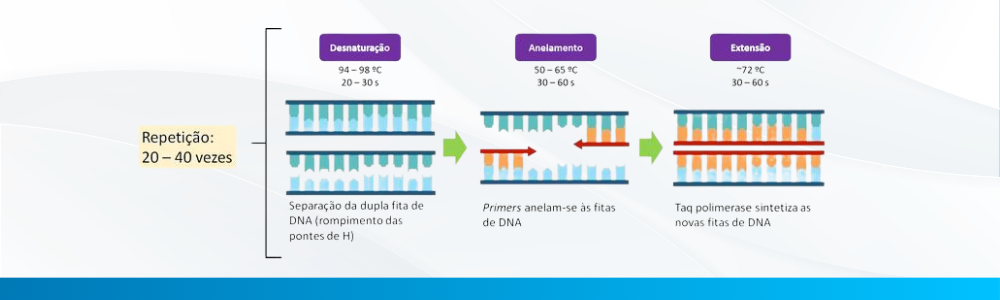
 A reação de PCR ocorre, em repetidos ciclos, com alterações de temperatura e de tempo, em um equipamento chamado termociclador, uma vez que cada etapa da reação necessita de uma temperatura específica
No início da reação, a molécula original, fita dupla, do DNA Template é submetida à alta temperatura, para que haja a separação das ligações dupla fita umas das outras, por meio da quebra das ligações de hidrogênio. Essa etapa é chamada de desnaturação. A temperatura de desnaturação na reação de PCR é de aproximadamente 94-98°C, e a duração desta etapa é de 20 a 30 segundos.
Em seguida, ocorre redução da temperatura, permitindo assim que os primers se liguem às suas sequências específicas na fita simples de DNA. Esta é a etapa de anelamento, que tem duração média de 20 a 60 segundos. A temperatura exata nesta etapa depende do comprimento e da composição dos primers (percentual de cada base nitrogenada), sendo aproximadamente de 50-65°C.
A próxima etapa da PCR é a extensão, cuja duração depende do comprimento do fragmento de DNA amplificado, mas que ocorre geralmente de 30 segundos a alguns minutos. E a temperatura de extensão fica, aproximadamente, entre 68-75°C, dependendo de qual DNA polimerase é utilizada. Nesta etapa, a DNA polimerase sintetiza novas cadeias de DNA, adicionando nucleotídeos complementares aos primers. A DNA polimerase vai se movendo ao longo do DNA simples, estendendo a nova cadeia de DNA, resultando na formação de uma cópia complementar do DNA alvo.
Essas três etapas se repetem em torno de 20 a 40 ciclos, normalmente, amplificando exponencialmente a quantidade do DNA alvo, permitindo a produção de milhões de cópias do segmento de interesse ao final dos ciclos.
A reação de PCR ocorre, em repetidos ciclos, com alterações de temperatura e de tempo, em um equipamento chamado termociclador, uma vez que cada etapa da reação necessita de uma temperatura específica
No início da reação, a molécula original, fita dupla, do DNA Template é submetida à alta temperatura, para que haja a separação das ligações dupla fita umas das outras, por meio da quebra das ligações de hidrogênio. Essa etapa é chamada de desnaturação. A temperatura de desnaturação na reação de PCR é de aproximadamente 94-98°C, e a duração desta etapa é de 20 a 30 segundos.
Em seguida, ocorre redução da temperatura, permitindo assim que os primers se liguem às suas sequências específicas na fita simples de DNA. Esta é a etapa de anelamento, que tem duração média de 20 a 60 segundos. A temperatura exata nesta etapa depende do comprimento e da composição dos primers (percentual de cada base nitrogenada), sendo aproximadamente de 50-65°C.
A próxima etapa da PCR é a extensão, cuja duração depende do comprimento do fragmento de DNA amplificado, mas que ocorre geralmente de 30 segundos a alguns minutos. E a temperatura de extensão fica, aproximadamente, entre 68-75°C, dependendo de qual DNA polimerase é utilizada. Nesta etapa, a DNA polimerase sintetiza novas cadeias de DNA, adicionando nucleotídeos complementares aos primers. A DNA polimerase vai se movendo ao longo do DNA simples, estendendo a nova cadeia de DNA, resultando na formação de uma cópia complementar do DNA alvo.
Essas três etapas se repetem em torno de 20 a 40 ciclos, normalmente, amplificando exponencialmente a quantidade do DNA alvo, permitindo a produção de milhões de cópias do segmento de interesse ao final dos ciclos.
 A técnica de PCR é muito sensível e específica, permitindo a amplificação de DNA a partir de uma quantidade mínima de material genético, o que favorece sua aplicação em diversas áreas, como na pesquisa básica e aplicada, em estudos de identificação e variabilidade genética, análises forenses, diagnósticos de doenças infecciosas, controle de qualidade industrial e engenharia genética, dentre outros. É uma técnica passível de realização em poucas horas, sendo mais rápida que métodos mais tradicionais, acelerando assim os processos de análises, pesquisas e diagnósticos. Com os avanços científicos das últimas décadas e uma ampla disponibilidade de reagentes e equipamentos, o custo da PCR reduziu consideravelmente, tornando-a bastante acessível. Isso tem possibilitou descobertas importantes em genética, como a identificação de genes associados a doenças, a realização de estudos detalhados sobre a estrutura e função dos genes e mapeamento genético.
A PCR transformou a biologia molecular, abrindo portas para uma nova era na pesquisa genética de diversas áreas e nas aplicações clínicas. E depois dela, o mundo da ciência nunca mais foi o mesmo.
Referência bibliográfica
Mullis, K. B., & Oliver, J. W. (1985). The polymerase chain reaction: a method for detecting specific sequences of DNA. BioTechniques, 3(4), 258-264.
Mullis, K. B., & Olsson, C. (1986). A rapid and sensitive method for detecting specific sequences of DNA. BioTechniques, 4(1), 32-39.
Mullis, K. B., & Faloona, F. (1987). Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods in Enzymology, 155, 335-350.
A técnica de PCR é muito sensível e específica, permitindo a amplificação de DNA a partir de uma quantidade mínima de material genético, o que favorece sua aplicação em diversas áreas, como na pesquisa básica e aplicada, em estudos de identificação e variabilidade genética, análises forenses, diagnósticos de doenças infecciosas, controle de qualidade industrial e engenharia genética, dentre outros. É uma técnica passível de realização em poucas horas, sendo mais rápida que métodos mais tradicionais, acelerando assim os processos de análises, pesquisas e diagnósticos. Com os avanços científicos das últimas décadas e uma ampla disponibilidade de reagentes e equipamentos, o custo da PCR reduziu consideravelmente, tornando-a bastante acessível. Isso tem possibilitou descobertas importantes em genética, como a identificação de genes associados a doenças, a realização de estudos detalhados sobre a estrutura e função dos genes e mapeamento genético.
A PCR transformou a biologia molecular, abrindo portas para uma nova era na pesquisa genética de diversas áreas e nas aplicações clínicas. E depois dela, o mundo da ciência nunca mais foi o mesmo.
Referência bibliográfica
Mullis, K. B., & Oliver, J. W. (1985). The polymerase chain reaction: a method for detecting specific sequences of DNA. BioTechniques, 3(4), 258-264.
Mullis, K. B., & Olsson, C. (1986). A rapid and sensitive method for detecting specific sequences of DNA. BioTechniques, 4(1), 32-39.
Mullis, K. B., & Faloona, F. (1987). Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods in Enzymology, 155, 335-350.

 Para a realização da PCR, alguns componentes obrigatórios devem integrar a reação:
DNA Template: Se trata do modelo de DNA, ou a amostra de DNA que contém a sequência alvo.
DNA Polymerase: Enzima que sintetiza novas fitas de DNA, complementares à sequência alvo. É uma enzima altamente resistente ao calor.
Primers: Sequências curtas de DNA de fita simples que são complementares à sequência alvo. A polimerase começa a sintetizar a nova fita de DNA a partir do final do primer.
Nucleotídeos, ou desoxinucleotídeos trifosfatos (dNTPs): São, essencialmente, “blocos de construção” para novas cadeias de DNA. Cada dNTP é composto por uma base nitrogenada, um açúcar e um grupos fosfato. As moléculas de DNA contêm quatro diferentes bases nitrogenadas que compõem um desoxinucleotídeo trifosfato: Adenina (A), Timina (T), Citosina (C) e Guanina (G). Já as moléculas de RNA são compostas pelas bases nitrogenadas: Adenina (A), Uracila (U), Citosina (C) e Guanina (G). São as bases nitrogenadas, presentes nos nucleotídeos, que codificam a informação genética, uma vez que é a sequência de bases ao longo da molécula de DNA e RNA que determina a informação genética.
Cloreto de magnésio (MgCl₂): Atua como cofator essencial da atividade da DNA Polimerase, além de influenciar na estabilização dos nucleotídeos e na especificidade da amplificação do alvo na reação.
Para a realização da PCR, alguns componentes obrigatórios devem integrar a reação:
DNA Template: Se trata do modelo de DNA, ou a amostra de DNA que contém a sequência alvo.
DNA Polymerase: Enzima que sintetiza novas fitas de DNA, complementares à sequência alvo. É uma enzima altamente resistente ao calor.
Primers: Sequências curtas de DNA de fita simples que são complementares à sequência alvo. A polimerase começa a sintetizar a nova fita de DNA a partir do final do primer.
Nucleotídeos, ou desoxinucleotídeos trifosfatos (dNTPs): São, essencialmente, “blocos de construção” para novas cadeias de DNA. Cada dNTP é composto por uma base nitrogenada, um açúcar e um grupos fosfato. As moléculas de DNA contêm quatro diferentes bases nitrogenadas que compõem um desoxinucleotídeo trifosfato: Adenina (A), Timina (T), Citosina (C) e Guanina (G). Já as moléculas de RNA são compostas pelas bases nitrogenadas: Adenina (A), Uracila (U), Citosina (C) e Guanina (G). São as bases nitrogenadas, presentes nos nucleotídeos, que codificam a informação genética, uma vez que é a sequência de bases ao longo da molécula de DNA e RNA que determina a informação genética.
Cloreto de magnésio (MgCl₂): Atua como cofator essencial da atividade da DNA Polimerase, além de influenciar na estabilização dos nucleotídeos e na especificidade da amplificação do alvo na reação.
 A reação de PCR ocorre, em repetidos ciclos, com alterações de temperatura e de tempo, em um equipamento chamado termociclador, uma vez que cada etapa da reação necessita de uma temperatura específica
No início da reação, a molécula original, fita dupla, do DNA Template é submetida à alta temperatura, para que haja a separação das ligações dupla fita umas das outras, por meio da quebra das ligações de hidrogênio. Essa etapa é chamada de desnaturação. A temperatura de desnaturação na reação de PCR é de aproximadamente 94-98°C, e a duração desta etapa é de 20 a 30 segundos.
Em seguida, ocorre redução da temperatura, permitindo assim que os primers se liguem às suas sequências específicas na fita simples de DNA. Esta é a etapa de anelamento, que tem duração média de 20 a 60 segundos. A temperatura exata nesta etapa depende do comprimento e da composição dos primers (percentual de cada base nitrogenada), sendo aproximadamente de 50-65°C.
A próxima etapa da PCR é a extensão, cuja duração depende do comprimento do fragmento de DNA amplificado, mas que ocorre geralmente de 30 segundos a alguns minutos. E a temperatura de extensão fica, aproximadamente, entre 68-75°C, dependendo de qual DNA polimerase é utilizada. Nesta etapa, a DNA polimerase sintetiza novas cadeias de DNA, adicionando nucleotídeos complementares aos primers. A DNA polimerase vai se movendo ao longo do DNA simples, estendendo a nova cadeia de DNA, resultando na formação de uma cópia complementar do DNA alvo.
Essas três etapas se repetem em torno de 20 a 40 ciclos, normalmente, amplificando exponencialmente a quantidade do DNA alvo, permitindo a produção de milhões de cópias do segmento de interesse ao final dos ciclos.
A reação de PCR ocorre, em repetidos ciclos, com alterações de temperatura e de tempo, em um equipamento chamado termociclador, uma vez que cada etapa da reação necessita de uma temperatura específica
No início da reação, a molécula original, fita dupla, do DNA Template é submetida à alta temperatura, para que haja a separação das ligações dupla fita umas das outras, por meio da quebra das ligações de hidrogênio. Essa etapa é chamada de desnaturação. A temperatura de desnaturação na reação de PCR é de aproximadamente 94-98°C, e a duração desta etapa é de 20 a 30 segundos.
Em seguida, ocorre redução da temperatura, permitindo assim que os primers se liguem às suas sequências específicas na fita simples de DNA. Esta é a etapa de anelamento, que tem duração média de 20 a 60 segundos. A temperatura exata nesta etapa depende do comprimento e da composição dos primers (percentual de cada base nitrogenada), sendo aproximadamente de 50-65°C.
A próxima etapa da PCR é a extensão, cuja duração depende do comprimento do fragmento de DNA amplificado, mas que ocorre geralmente de 30 segundos a alguns minutos. E a temperatura de extensão fica, aproximadamente, entre 68-75°C, dependendo de qual DNA polimerase é utilizada. Nesta etapa, a DNA polimerase sintetiza novas cadeias de DNA, adicionando nucleotídeos complementares aos primers. A DNA polimerase vai se movendo ao longo do DNA simples, estendendo a nova cadeia de DNA, resultando na formação de uma cópia complementar do DNA alvo.
Essas três etapas se repetem em torno de 20 a 40 ciclos, normalmente, amplificando exponencialmente a quantidade do DNA alvo, permitindo a produção de milhões de cópias do segmento de interesse ao final dos ciclos.
 A técnica de PCR é muito sensível e específica, permitindo a amplificação de DNA a partir de uma quantidade mínima de material genético, o que favorece sua aplicação em diversas áreas, como na pesquisa básica e aplicada, em estudos de identificação e variabilidade genética, análises forenses, diagnósticos de doenças infecciosas, controle de qualidade industrial e engenharia genética, dentre outros. É uma técnica passível de realização em poucas horas, sendo mais rápida que métodos mais tradicionais, acelerando assim os processos de análises, pesquisas e diagnósticos. Com os avanços científicos das últimas décadas e uma ampla disponibilidade de reagentes e equipamentos, o custo da PCR reduziu consideravelmente, tornando-a bastante acessível. Isso tem possibilitou descobertas importantes em genética, como a identificação de genes associados a doenças, a realização de estudos detalhados sobre a estrutura e função dos genes e mapeamento genético.
A PCR transformou a biologia molecular, abrindo portas para uma nova era na pesquisa genética de diversas áreas e nas aplicações clínicas. E depois dela, o mundo da ciência nunca mais foi o mesmo.
Referência bibliográfica
Mullis, K. B., & Oliver, J. W. (1985). The polymerase chain reaction: a method for detecting specific sequences of DNA. BioTechniques, 3(4), 258-264.
Mullis, K. B., & Olsson, C. (1986). A rapid and sensitive method for detecting specific sequences of DNA. BioTechniques, 4(1), 32-39.
Mullis, K. B., & Faloona, F. (1987). Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods in Enzymology, 155, 335-350.
A técnica de PCR é muito sensível e específica, permitindo a amplificação de DNA a partir de uma quantidade mínima de material genético, o que favorece sua aplicação em diversas áreas, como na pesquisa básica e aplicada, em estudos de identificação e variabilidade genética, análises forenses, diagnósticos de doenças infecciosas, controle de qualidade industrial e engenharia genética, dentre outros. É uma técnica passível de realização em poucas horas, sendo mais rápida que métodos mais tradicionais, acelerando assim os processos de análises, pesquisas e diagnósticos. Com os avanços científicos das últimas décadas e uma ampla disponibilidade de reagentes e equipamentos, o custo da PCR reduziu consideravelmente, tornando-a bastante acessível. Isso tem possibilitou descobertas importantes em genética, como a identificação de genes associados a doenças, a realização de estudos detalhados sobre a estrutura e função dos genes e mapeamento genético.
A PCR transformou a biologia molecular, abrindo portas para uma nova era na pesquisa genética de diversas áreas e nas aplicações clínicas. E depois dela, o mundo da ciência nunca mais foi o mesmo.
Referência bibliográfica
Mullis, K. B., & Oliver, J. W. (1985). The polymerase chain reaction: a method for detecting specific sequences of DNA. BioTechniques, 3(4), 258-264.
Mullis, K. B., & Olsson, C. (1986). A rapid and sensitive method for detecting specific sequences of DNA. BioTechniques, 4(1), 32-39.
Mullis, K. B., & Faloona, F. (1987). Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods in Enzymology, 155, 335-350.



