06 de Fevereiro de 2025
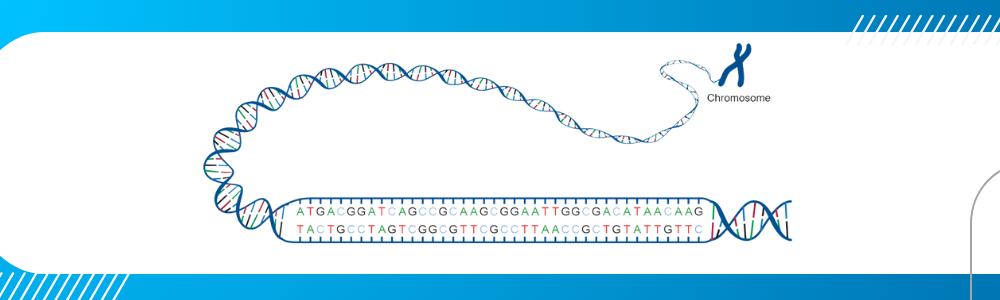 As informações genéticas de um organismo estão armazenadas em seu genoma. O genoma é o conjunto total de DNA presente em cada célula, e é no DNA (e a partir do DNA) que toda informação genética é armazenada, transmitida e expressa.
A estrutura da molécula de DNA foi elucidada na década de 50 e esta descoberta desencadeou inúmeras possibilidades, em muitas áreas das Ciências Biológicas.
As informações genéticas de um organismo estão armazenadas em seu genoma. O genoma é o conjunto total de DNA presente em cada célula, e é no DNA (e a partir do DNA) que toda informação genética é armazenada, transmitida e expressa.
A estrutura da molécula de DNA foi elucidada na década de 50 e esta descoberta desencadeou inúmeras possibilidades, em muitas áreas das Ciências Biológicas.
 Após a descoberta da estrutura do DNA, os cientistas passaram a investigar o fato de que, de algum modo, esta molécula continha todos os códigos para a produção das proteínas, funcionando como uma espécie de “livro de receitas”, com as instruções para a formação de qualquer organismo vivo. Porém, até então, o desafio era: como acessar e decifrar o conteúdo desse “livro de receitas”?
Após a confirmação da existência do código genético, as pesquisas científicas nessa área trabalharam para encontrar uma maneira de extrair esse código em forma de dados. Era necessário ter acesso às informações (às letrinhas) desse fantástico livro de receitas.
No âmbito da biologia molecular, a década de 70 foi marcada por avanços significativos, como a clonagem de genes e a PCR (Reação em Cadeia da Polimerase) que passaram a ser amplamente utilizadas na preparação de bibliotecas de DNA. Também nessa década foram desenvolvidos computadores mais poderosos e software mais adequados para processar dados de bioinformática e, partir desse contexto, surgiram as abordagens de sequenciamento. Tais avanços abriram portas para um entendimento mais aprofundado da diversidade e funcionamento de diversos organismos, e de como os organismos respondem às alterações em seu código genético.
______________________________
Sequenciamento Genético é a técnica que identifica uma sequência (a ordem) de ácidos nucleicos em determinada região do material genético de uma determinada amostra biológica.
As primeiras ações relacionadas à sequenciamento genético basearam-se em dois métodos, e se agrupam em uma classificação conhecida como Sequenciamento de Primeira Geração: 1Degradação Química - Método Maxam e Gilbert; e 2Interrupção da Cadeia com dideoxinucleotídeos (ddNTPs) - Método Sanger.
O Método de Maxam e Gilbert foi a abordagem pioneira dentre os métodos de sequenciamento, no início dos anos 70 e constitui, basicamente, na utilização de uma marcação do DNA alvo a ser sequenciado com uma molécula de fósforo radioativo (P32). Porém, dentre os métodos de Primeira Geração, o Sequenciamento Sanger se destacou como o mais utilizado uma vez que, a partir do sequenciamento por degradação química, de Maxam e Gilbert, Frederick Sanger propôs melhorias significativas à técnica de sequenciamento genético, tornando-a mais viável e acessível.
Os primeiros passos na tentativa de decifrar o código genético: SEQUENCIAMENTO SANGER
O Sequenciamento Sanger foi a técnica que deu início ao sequenciamento massivo de DNA e foi a tecnologia que possibilitou, posteriormente, a realização do Projeto Genoma Humano, em 1991. Este projeto que envolveu cientistas do mundo todo publicou, em 2001, o primeiro rascunho do genoma humano, graças às técnicas Sanger e, simultaneamente neste período, graças ao desenvolvimento de técnicas inovadoras de sequenciamento massivo. A partir deste projeto, foi possível a leitura de todo o material genético dos humanos e isso abriu portas para inúmeras inovações científicas e tecnológicas.
A técnica de Sequenciamento Sanger, também chamada de dedesoxi, utiliza uma enzima DNA polimerase para a síntese de cadeias de DNA de diferentes comprimentos, onde uma fita de DNA servirá de molde para a fita da qual se deseja investigar a sequência. Deste modo, alguns componentes são fundamentais para uma reação de sequenciamento Sanger: 1Molécula de DNA, o DNA molde da sequência que se deseja conhecer; 2Os quatro nucleotídeos (dNTPs): Adenina (dATP), Timina (dTTP), Citosina (dCTP) e Guanina (dGTP); 3Didesoxinucleotídeos, que são um tipo especial de nucleotídeo (chamados também de ddNTPs), porém com modificações: são modificados com agentes fluorescentes chamados fluoróforos (moléculas que emitem luminescência), e terminadores da cadeia, 4Sequência iniciadora (primer); 5Enzima polimerase.
No processo de sequenciamento Sanger, o primer é o ponto de partida da reação, ligando-se a uma região complementar na sequência do DNA alvo, iniciando a síntese de uma nova fita. A DNA polimerase é a enzima responsável pela adição de novos nucleotídeos à nova fita. Os fragmentos, de diferentes tamanhos são sequenciados e identificados a partir da fluorescência emitida pelo último nucleotídeo adicionado.
O Sequenciamento Sanger analisa fragmentos de DNA de até 800pb. Esta técnica utiliza nucleotídeos modificados com moléculas que emitem luminescência, chamadas fluoróforos, e produz fragmentos de diferentes tamanhos da sequência do DNA alvo de interesse. Esses fragmentos produzidos são separados por tamanhos e as bases finais de cada cópia são identificadas pela fluorescência emitida. Isso permite que a sequência do DNA de interesse seja determinada.
Após a descoberta da estrutura do DNA, os cientistas passaram a investigar o fato de que, de algum modo, esta molécula continha todos os códigos para a produção das proteínas, funcionando como uma espécie de “livro de receitas”, com as instruções para a formação de qualquer organismo vivo. Porém, até então, o desafio era: como acessar e decifrar o conteúdo desse “livro de receitas”?
Após a confirmação da existência do código genético, as pesquisas científicas nessa área trabalharam para encontrar uma maneira de extrair esse código em forma de dados. Era necessário ter acesso às informações (às letrinhas) desse fantástico livro de receitas.
No âmbito da biologia molecular, a década de 70 foi marcada por avanços significativos, como a clonagem de genes e a PCR (Reação em Cadeia da Polimerase) que passaram a ser amplamente utilizadas na preparação de bibliotecas de DNA. Também nessa década foram desenvolvidos computadores mais poderosos e software mais adequados para processar dados de bioinformática e, partir desse contexto, surgiram as abordagens de sequenciamento. Tais avanços abriram portas para um entendimento mais aprofundado da diversidade e funcionamento de diversos organismos, e de como os organismos respondem às alterações em seu código genético.
______________________________
Sequenciamento Genético é a técnica que identifica uma sequência (a ordem) de ácidos nucleicos em determinada região do material genético de uma determinada amostra biológica.
As primeiras ações relacionadas à sequenciamento genético basearam-se em dois métodos, e se agrupam em uma classificação conhecida como Sequenciamento de Primeira Geração: 1Degradação Química - Método Maxam e Gilbert; e 2Interrupção da Cadeia com dideoxinucleotídeos (ddNTPs) - Método Sanger.
O Método de Maxam e Gilbert foi a abordagem pioneira dentre os métodos de sequenciamento, no início dos anos 70 e constitui, basicamente, na utilização de uma marcação do DNA alvo a ser sequenciado com uma molécula de fósforo radioativo (P32). Porém, dentre os métodos de Primeira Geração, o Sequenciamento Sanger se destacou como o mais utilizado uma vez que, a partir do sequenciamento por degradação química, de Maxam e Gilbert, Frederick Sanger propôs melhorias significativas à técnica de sequenciamento genético, tornando-a mais viável e acessível.
Os primeiros passos na tentativa de decifrar o código genético: SEQUENCIAMENTO SANGER
O Sequenciamento Sanger foi a técnica que deu início ao sequenciamento massivo de DNA e foi a tecnologia que possibilitou, posteriormente, a realização do Projeto Genoma Humano, em 1991. Este projeto que envolveu cientistas do mundo todo publicou, em 2001, o primeiro rascunho do genoma humano, graças às técnicas Sanger e, simultaneamente neste período, graças ao desenvolvimento de técnicas inovadoras de sequenciamento massivo. A partir deste projeto, foi possível a leitura de todo o material genético dos humanos e isso abriu portas para inúmeras inovações científicas e tecnológicas.
A técnica de Sequenciamento Sanger, também chamada de dedesoxi, utiliza uma enzima DNA polimerase para a síntese de cadeias de DNA de diferentes comprimentos, onde uma fita de DNA servirá de molde para a fita da qual se deseja investigar a sequência. Deste modo, alguns componentes são fundamentais para uma reação de sequenciamento Sanger: 1Molécula de DNA, o DNA molde da sequência que se deseja conhecer; 2Os quatro nucleotídeos (dNTPs): Adenina (dATP), Timina (dTTP), Citosina (dCTP) e Guanina (dGTP); 3Didesoxinucleotídeos, que são um tipo especial de nucleotídeo (chamados também de ddNTPs), porém com modificações: são modificados com agentes fluorescentes chamados fluoróforos (moléculas que emitem luminescência), e terminadores da cadeia, 4Sequência iniciadora (primer); 5Enzima polimerase.
No processo de sequenciamento Sanger, o primer é o ponto de partida da reação, ligando-se a uma região complementar na sequência do DNA alvo, iniciando a síntese de uma nova fita. A DNA polimerase é a enzima responsável pela adição de novos nucleotídeos à nova fita. Os fragmentos, de diferentes tamanhos são sequenciados e identificados a partir da fluorescência emitida pelo último nucleotídeo adicionado.
O Sequenciamento Sanger analisa fragmentos de DNA de até 800pb. Esta técnica utiliza nucleotídeos modificados com moléculas que emitem luminescência, chamadas fluoróforos, e produz fragmentos de diferentes tamanhos da sequência do DNA alvo de interesse. Esses fragmentos produzidos são separados por tamanhos e as bases finais de cada cópia são identificadas pela fluorescência emitida. Isso permite que a sequência do DNA de interesse seja determinada.
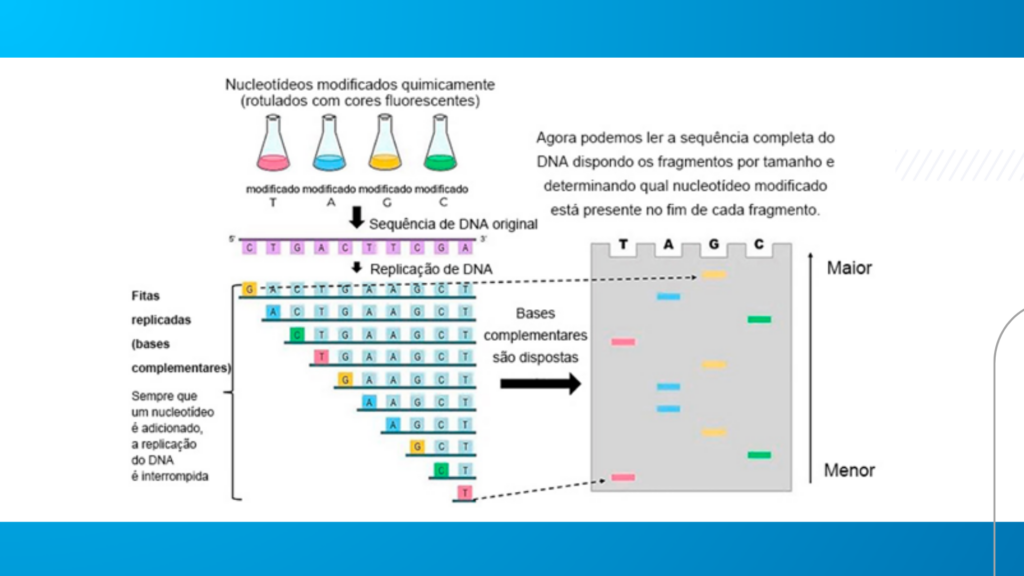 Representação da técnica de sequenciamento Sanger.
Esta técnica de sequenciamento foi tão pioneira que rendeu ao cientista Frederick Sanger o Prêmio Nobel de Química, em 1980. E os avanços técnicos nas décadas seguintes, aperfeiçoaram ainda mais o processo de sequenciamento Sanger, tornando-a automatizada.
A principal vantagem do método de sequenciamento Sanger, em relação aos métodos subsequentes, é o maior tamanho dos fragmentos (reads) gerados, e a precisão da base de dados gerada. O sequenciamento Sanger foi base para os métodos subsequentes, conhecidos como de Segunda Geração, que tiveram início com a primeira técnica NGS, e que diminuíram consideravelmente os custos e o tempo dos sequenciamentos de DNA.
Sequenciamento de Nova Geração - NGS (Next-Generation Sequencing)
Sequenciamento NGS é o termo conhecido para o conjunto de técnicas modernas de sequenciamento, conhecido como sequenciamento massivo (bilhões de fragmentos de DNA) em paralelo (simultaneamente) ou de alto rendimento. Esta técnica permite a leitura rápida e em larga escala de fragmentos de DNA e pode ser utilizada para análises de regiões/genes específicas, exomas completos ou painéis moleculares (análise simultânea de vários alvos). Portanto, o sequenciamento de nova geração é uma alternativa poderosa para estudos de genômica estrutural e funcional.
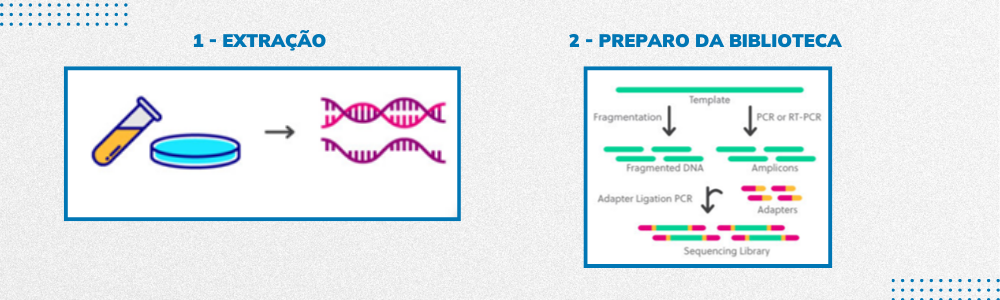
Inicialmente a amostra de DNA/RNA extraído é fragmentada em alíquotas menores. Esses fragmentos são ligados à adaptadores (sequências conhecidas em suas extremidades) e em seguida, amplificados.
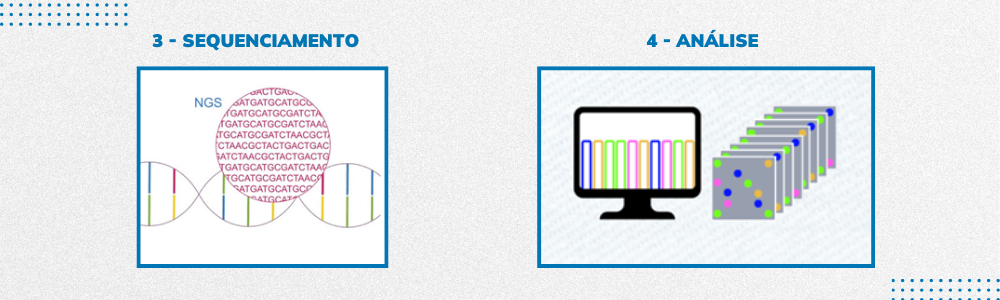
Em uma plataforma NGS, os fragmentos são sequenciados simultaneamente, produzindo um grande volume de dados. A sequência de nucleotídeos de cada fragmento é identificada e alinhada, de modo a gerar uma leitura completa da região de interesse ou do genoma completo.
Posteriormente, com ferramentas computacionais de Bioinformática, esses dados são analisados e, tal análise permite a reconstrução da sequência de DNA original ou a identificação de variações genéticas que se busca investigar.
A técnica NGS se diferencia por sua capacidade de sequenciar milhões de fragmentos em paralelo, o que caracteriza eficiência e rapidez ao processo.
Representação da técnica de sequenciamento Sanger.
Esta técnica de sequenciamento foi tão pioneira que rendeu ao cientista Frederick Sanger o Prêmio Nobel de Química, em 1980. E os avanços técnicos nas décadas seguintes, aperfeiçoaram ainda mais o processo de sequenciamento Sanger, tornando-a automatizada.
A principal vantagem do método de sequenciamento Sanger, em relação aos métodos subsequentes, é o maior tamanho dos fragmentos (reads) gerados, e a precisão da base de dados gerada. O sequenciamento Sanger foi base para os métodos subsequentes, conhecidos como de Segunda Geração, que tiveram início com a primeira técnica NGS, e que diminuíram consideravelmente os custos e o tempo dos sequenciamentos de DNA.
Sequenciamento de Nova Geração - NGS (Next-Generation Sequencing)
Sequenciamento NGS é o termo conhecido para o conjunto de técnicas modernas de sequenciamento, conhecido como sequenciamento massivo (bilhões de fragmentos de DNA) em paralelo (simultaneamente) ou de alto rendimento. Esta técnica permite a leitura rápida e em larga escala de fragmentos de DNA e pode ser utilizada para análises de regiões/genes específicas, exomas completos ou painéis moleculares (análise simultânea de vários alvos). Portanto, o sequenciamento de nova geração é uma alternativa poderosa para estudos de genômica estrutural e funcional.
Inicialmente a amostra de DNA/RNA extraído é fragmentada em alíquotas menores. Esses fragmentos são ligados à adaptadores (sequências conhecidas em suas extremidades) e em seguida, amplificados.
Em uma plataforma NGS, os fragmentos são sequenciados simultaneamente, produzindo um grande volume de dados. A sequência de nucleotídeos de cada fragmento é identificada e alinhada, de modo a gerar uma leitura completa da região de interesse ou do genoma completo.
Posteriormente, com ferramentas computacionais de Bioinformática, esses dados são analisados e, tal análise permite a reconstrução da sequência de DNA original ou a identificação de variações genéticas que se busca investigar.
A técnica NGS se diferencia por sua capacidade de sequenciar milhões de fragmentos em paralelo, o que caracteriza eficiência e rapidez ao processo.
Etapas de um sequenciamento NGS:
 As técnicas de sequenciamento NGS podem ser aplicadas em diversas áreas de estudo, como no contexto médico, por exemplo: na identificação de mutações patogênicas, diagnóstico de doenças genéticas (como doenças raras e pesquisas em tumores), dentre outras linhas de pesquisa médicas. Analisando as aplicações no âmbito da biodiversidade, observa-se diversas possibilidades na utilização de NGS, tanto na área da saúde como em pesquisas ecológicas como: microbioma intestinal de humanos e outros animais, análises de microbioma de solo, água, ar, estudos de variabilidade populacional de diversos organismos, em escalas geográficas e temporais.
A verdade é que, independentemente da área, do objetivo e do objeto de estudo, há sempre um vasto nicho de aplicações para técnicas de NGS.
Desde o desenvolvimento das primeiras técnicas de NGS, que hoje são conhecidas como de Segunda geração, muita evolução tecnológica ocorreu, no âmbito do desenvolvimento de equipamentos e software de processamento e análise de dados. Nos últimos 20 anos, já foram desenvolvidos sequenciadores de terceira e quarta geração. Todas as técnicas são aplicáveis, de qualquer modo, no sequenciamento de DNA, havendo a possibilidade de análise indireta também de RNA (RNA-Seq) e proteínas.
Há um vasto mundo de possibilidades e recursos (de máquinas e software), para qualquer tipo de análise relacionada à Biologia Molecular e Genética. A cada nova geração de técnicas de sequenciamento, foram surgindo equipamentos e inovações, cada vez mais aptos a atender demandas específicas. Segue abaixo alguns deles:
Técnicas de Sequenciamento de Segunda Geração mais populares:
As técnicas de sequenciamento NGS podem ser aplicadas em diversas áreas de estudo, como no contexto médico, por exemplo: na identificação de mutações patogênicas, diagnóstico de doenças genéticas (como doenças raras e pesquisas em tumores), dentre outras linhas de pesquisa médicas. Analisando as aplicações no âmbito da biodiversidade, observa-se diversas possibilidades na utilização de NGS, tanto na área da saúde como em pesquisas ecológicas como: microbioma intestinal de humanos e outros animais, análises de microbioma de solo, água, ar, estudos de variabilidade populacional de diversos organismos, em escalas geográficas e temporais.
A verdade é que, independentemente da área, do objetivo e do objeto de estudo, há sempre um vasto nicho de aplicações para técnicas de NGS.
Desde o desenvolvimento das primeiras técnicas de NGS, que hoje são conhecidas como de Segunda geração, muita evolução tecnológica ocorreu, no âmbito do desenvolvimento de equipamentos e software de processamento e análise de dados. Nos últimos 20 anos, já foram desenvolvidos sequenciadores de terceira e quarta geração. Todas as técnicas são aplicáveis, de qualquer modo, no sequenciamento de DNA, havendo a possibilidade de análise indireta também de RNA (RNA-Seq) e proteínas.
Há um vasto mundo de possibilidades e recursos (de máquinas e software), para qualquer tipo de análise relacionada à Biologia Molecular e Genética. A cada nova geração de técnicas de sequenciamento, foram surgindo equipamentos e inovações, cada vez mais aptos a atender demandas específicas. Segue abaixo alguns deles:
Técnicas de Sequenciamento de Segunda Geração mais populares:
- Amplicon
- Hibridação e captura (PCR-free)
- Sequenciamento Illumina: Sequenciamento por síntese
- Ion Torrent
- Nanopore
- Sequenciamento PacBio (SMRT)
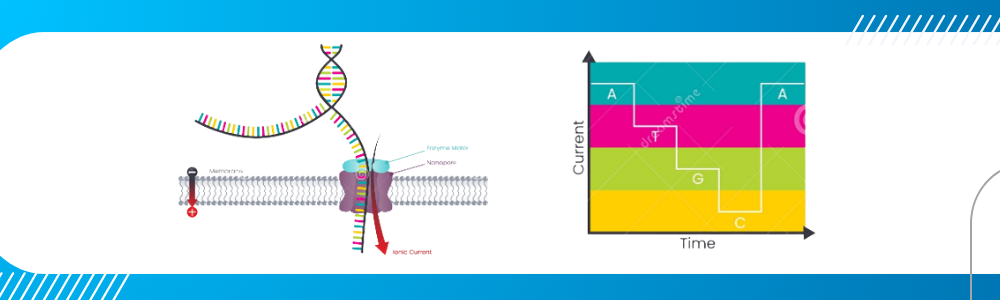 Representação da técnica de sequenciamento Nanopore.
Representação da técnica de sequenciamento Nanopore.
Sequenciamento de Quarta Geração: in situ sequencing
Esta geração de sequenciamento tem por objetivo localizar e compreender o comportamento de moléculas, como RNAs e proteínas, porém a nível celular, ainda dentro da célula. Sequenciadores de quarta geração constituem uma união das técnicas de segunda e terceiras gerações. Até a terceira geração, não havia a possibilidade de se localizar com exatidão as moléculas dentro das células, pois as análises são realizadas com material genética (DNA, RNA, proteínas) extraído de algum organismo e contendo várias células juntas. No sequenciamento de quarta geração, as análises de expressão gênica são capazes de identificar e localizar cada RNA, o que auxilia no estudo funcional dessas moléculas e da própria célula. As técnicas da quarta geração também possibilitam a realização de análises de complexos de proteínas, no que se refere à localização e função na célula. A identificação de variantes também é realizada, porém demanda um tempo maior de análise e curto mais elevado. O avanço das técnicas de sequenciamento, especialmente das de quarta geração terá um papel determinante em diversas áreas científicas, tanto voltadas para a saúde humana, como para diversas outras áreas, como ecologia, agricultura, pecuária. No que se refere à saúde humana, o sequenciamento de quarta geração será fundamental em estudos de terapia gênica, como alvos terapêuticos para o desenvolvimento de fármacos, em estudos sobre progressão de doenças degenerativas e tumores. Todos os esforços das últimas décadas, voltados para análises genéticas contribuirão, cada vez mais, para o avanço de diversas áreas científicas, especialmente para a área da saúde, garantindo melhor qualidade de vida e respostas para muitas perguntas que, até há tempo, eram indecifráveis. Referência bibliográfica Suzuki, Y. Advent of a new sequencing era: long-read and on-site sequencing. J Hum Genet 65, 1 (2020). https://doi.org/10.1038/s10038-019-0683-4 Tanimoto, I.M.F., Cressiot, B., Greive, S.J. et al. Focus on using nanopore technology for societal health, environmental, and energy challenges. Nano Res. 15, 9906–9920 (2022). https://doi.org/10.1007/s12274-022-4379-2 Watson, J., Crick, F. Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. Nature 171, 737–738 (1953). https://doi.org/10.1038/171737a0


